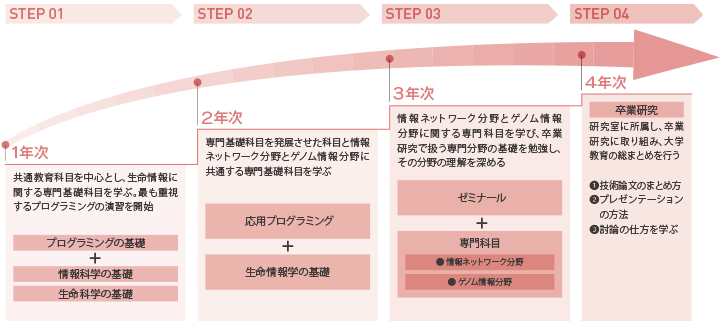
生命科学と情報科学の基礎及びプログラミング技術を修得し、4年次には研究室でゲノム情報や情報ネットワーク分野における問題を解決する卒業研究に取り組みます。実習とチームワークを重視しています。
ゼミナール

生命情報学科の学生は、3年次後期に研究室に仮配属になりゼミナールを受講します。受講者は事前に研究室を訪問して教官と話し、希望の研究室を選びます。当学科では各研究室6名以下の割り振りになっています。少人数制のジムで好きなメニューを選び、インストラクターの個別指導を受け、参加者が互いに声を掛け合って研究のための基礎的な筋肉をつけます。和やかな雰囲気の中で知識を血肉とすることができるのは、小さな大学が持つ大きなメリットです。
情報ネットワーク

日常利用している携帯電話、電子メール、ウェブページなどは、どのようにつながっているのでしょうか?データ送受信の仕組みを明らかにし、情報ネットワーク全体の展望を示します。また、機械同士の通信(Internet of Things: IoT) など、今後のネットワークの展開に伴い、生活がどのように変化していくのか考えていきます。
研究室
理論生物研究室 担当教員/本間 桂一

DNA配列などの生物関係の大量データの山をコンピュータにより選別し、病気の発症メカニズムなどの宝探しをしています。
テーマ例
- タンパク質の構造、配列、機能解析/ゲノム解析/DNAの変異と疾病の関係
学びのキーワード
- バイオインフォマティクス/ゲノム情報処理/データベース生物学、タンパク質/構造生物学/天然変性領域
生物情報化学研究室 担当教員/中村 建介
われわれ生物の姿や行動がどのようにして形作られているのかを知るために、ゲノムDNAの持つ「情報」を計算機で調べています。

テーマ例
- 次世代シーケンサによる塩基配列データの解析
- 分子軌道計算・分子力場計算・分子動力学計算などによる計算化学解析
- タンパク質の配列及び立体構造解析・基質との相互
学びのキーワード
- バイオインフォマティクス/微生物ゲノム解析/金属タンパク質/理論有機化学/次世代シーケンサ
タンパク質情報研究室 担当教員/福地 佐斗志
近年、生物学で得られるようになった大量のデータをコンピュータで解析し新しいことを発見したいと思い頑張っています。

テーマ例
- タンパク質構造・配列解析、ゲノム情報処理
- 生命情報データベース構築等
学びのキーワード
- バイオインフォマティックス/データベース生物学/プロテオーム情報処理
ネットワークセキュリティ研究室 担当教員/三河 賢治
パズルのような数学問題から、ネットワークセキュリティの技術開発、セキュリティを意識した情報システムの設計まで、はばの広いテーマを研究しています。
テーマ例
- 組合せ集合の列挙手法の設計
- パケット分類に適したデータ構造の開発
- セキュアな情報システムの設計
学びのキーワード
- アルゴリズム/ネットワークセキュリティ/情報システム設計
オミックス情報処理研究室 担当教員/蒔田 由布子
植物のゲノム配列や環境データを解析することで、生物と環境の相互作用を理解し、農業や環境保護への貢献を目指しています。

テーマ例
- 遺伝子の発現制御メカニズムの解明
- データベース構築
- 植物ゲノム科学おける環境と遺伝子制御
学びのキーワード
- 植物バイオインフォマティクス/データサイエンス/次世代シーケンサー
バイオインフォマティクス研究室 担当教員/佐藤 眞木彦
遺伝的アルゴリズムやサポートベクトルマシンなどの知的情報処理を中心に、ウェーブレット変換などとも組合わせた新たな適用分野を研究しています。

テーマ例
- バイオインフォマティクス、及びそのシステム
- 遺伝的アルゴリズムを適用した最適化問題
学びのキーワード
- バイオインフォマティクス/タンパク質の立体構造予測/遺伝的アルゴリズム/免疫アルゴリズム/最適化
システム生物学研究室 担当教員/関口 達也
酵素反応の解析を行っています。そのために必要なアプリの開発や、微生物の代謝過程を数式で表現する方法の研究を行っています。

テーマ例
- システム生物学実験研究者のための生化学反応シミュレータの設計・開発
学びのキーワード
- バイオインフォマティクス/コンピュータシミュレーション/生体生命システム情報学
理論計算機科学研究室 担当教員/遠山 宏明
コンピュータで問題を解くために用いるアルゴリズム(計算方法)とそれに要する計算の手間(計算量)に関する研究しています。

テーマ例
- クラスPとNP間の階層に関する研究
- 最適化問題に対する近似手法に関する研究
- 量子情報理論に関する基礎的研究
学びのキーワード
- 計算量理論/近似アルゴリズム/量子情報理論
生体分子ダイナミクス研究室 担当教員/優 乙石
蛋白質など生体分子の形や動きを分子レベルで調べています。コンピューターで分子をモデリングして、計算機によってその動きをシミュレーションしています。

テーマ例
- 蛋白質の形や動きを分子レベルで調べる
- 蛋白質と小さな分子(水分子や薬剤分子など)との相互作用を調べる
- 細胞内の特殊な環境が蛋白質の機能に与える影響を調べる
学びのキーワード
- 分子シミュレーション/蛋白質立体構造、ダイナミクス、相互作用/細胞環境
情報システム研究室 担当教員/冨澤 眞樹
拡張現実を利用したカード型教材、遊びながら学ぶという療育システムや小型コンピュータを使ったセンサーノードを開発しています。

テーマ例
- 情報システムの分析/設計/開発/評価(情報推薦システム、コンテンツマネジメントシステム、教育システム)、組込みシステム(マイクロプロセッサ応用、組込みソフトウェア)
学びのキーワード
- 計算機システム/情報ネットワーク/ソフトウェア/ウェブ情報学/学習支援システム
将来について
取得可能な資格
- ITパスポート試験
- 基本情報技術者試験
- 応用情報技術者試験
- 医療情報技師
卒業後の主な進路
- 本学または他大学の大学院進学
- 情報通信関連企業
- 製造関連企業(機械・電気・精密機器など)
- 金融機関
- 公務員 ほか